来源:DeepTech深科技
近日,北京大学化学与分子工程学院本科校友、美国威斯康星麦迪逊大学博士毕业生、目前正在斯坦福大学从事博士后研究的朱彦宇和所在团队研发出一种高时空分辨率的活细胞 DNA 成像技术 Oligo-LiveFISH。该技术基于 sgRNA 的序列特异性识别能力和定制化寡核苷酸库(oligo pool)设计而来。对于包括原代细胞在内的多种细胞类型中的重复基因组位点和非重复基因组位点,该技术能够对其进行成像和动态追踪,为染色质动态结构研究提供了一款全新工具。


基于 CRISPR 的动态 DNA 成像
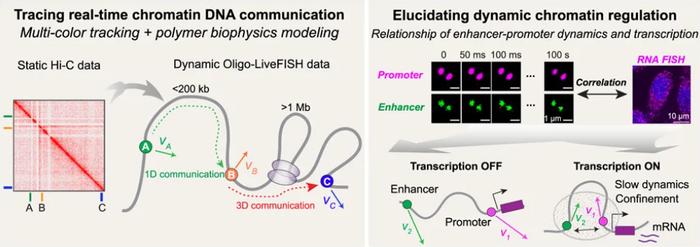
为了阐明 DNA 位点间的动态协同作用机制,研究团队提出了 DNA 通讯时间(DNA communication time)这一概念,即同一染色体上两个位点通过应力传递来感知彼此运动的时间延迟。基于本次 Oligo-LiveFISH 技术,研究团队针对不同 DNA 位点进行了不同时间尺度的动态追踪,进而提出两种 DNA 动态相互作用模式,它们分别是:一维顺式通讯(1D cis-communication)和三维反式通讯(3D trans-communication)。一维顺式通讯通过 DNA 聚合物链段的弹性行为来传递信息,它能主导小于 300kb 的短程位点的相互作用,其动力学特征符合经典 Rouse 模型的预测。三维反式通讯则显著存在大于 1Mb 的长程相互作用之中,其允许位点绕过 DNA 链,通过桥接复合物这一捷径在三维空间中加速通讯。在染色质动态调控中,这两种模式功能互补、协同作用。Oligo-LiveFISH 的单细胞动态数据不仅与 Hi-C 结果保持一致,同时还能提供传统测序技术或 FISH 技术无法捕捉的实时动态信息。
研究团队表示,在基因转录中,增强子-启动子的相互作用扮演重要角色。但是,增强子如何动态地与目标启动子相互作用?这种相互作用又是如何精准调控基因转录的?对于这两个问题,目前仍不明确。对于基因转录调控机制,本次研究团队研究了基因转录与其增强子-启动子动态行为之间的关联。以 FOS 基因为例,他们发现当基因转录的时候,其增强子和启动子之间的三维空间距离减小,约束性增加,并且运动变慢,形成一种更加稳定的相互作用。研究团队推测这可能是由于在转录过程中,转录因子和 RNA 聚合酶的募集增加了染色质微环境的拥挤程度。
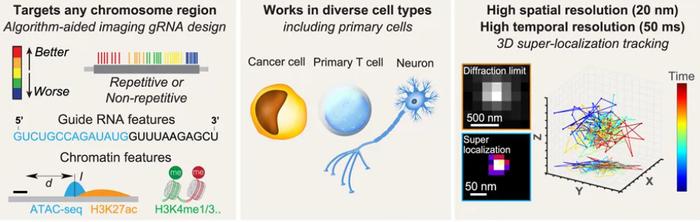
在机器学习的帮助之下,研究团队分析了多种 gRNA 参数和染色质特征对于成像效率的影响。他们发现 gRNA 探针数量和染色质可及性是影响 DNA 成像效果的核心因素,他们据此建立了“可视化不同细胞类型中的各种基因组位点”的 gRNA 设计原则。以此为原则,他们设计了针对不同染色体上的非重复基因组位点的探针,从而验证了 Oligo-LiveFISH 技术的普适性。为了方便用户使用,他们还开发了 Oligo-LiveFISH gRNA 探针设计平台(https://oligo-livefish.org/)。值得注意的是,尽管上述 gRNA 设计原则是基于 CRISPR 成像总结出来的,但是也有希望助力于基于 CRISPR 的干扰技术,比如 CRISPR 干扰/激活(CRISPRi/ CRISPRa)的 gRNA 设计。
审稿人认为,对于活细胞内非重复基因组位点的高时空分辨率成像来说,Oligo-LiveFISH 是非常重要的技术性突破,并认为将 Oligo-LiveFISH 用于针对之前难以成像的原代细胞进行染色体成像具有非常重要的意义。
在基础科研上,Oligo-LiveFISH 有望解答染色质三维结构、动态、与基因转录调控的因果关系这一关键科学问题。同时,还可用于观测包括 DNA 复制在内的许多重要的动态生命过程。
在潜在疾病研究上,Oligo-LiveFISH 作为一种活细胞 DNA 成像技术,可用于观测各个时间尺度下的多种细胞类型,从而有望在发育与疾病等领域实现突破性发现。比如,可以用其观测患者细胞分化过程中染色体的动态变化,检测患者与健康人细胞中的染色体动态差异进而揭示相关疾病的机理。
(来源:Cell)

“他山之石,可以攻玉”
在调控基因转录和细胞功能中,真核生物染色体的三维结构和动态变化起着重要作用。包括癌症、神经系统疾病、发育异常等疾病的发病机制,往往与三维基因组结构异常有关。比如,最近有研究表明,某些染色体区域三维结构的变化与肌萎缩侧索硬化症即渐冻症(ALS,Amyotrophic Lateral Sclerosis)等神经疾病有关,但对于它在功能上的具体作用机制仍不明确。
尽管传统成像方法比如荧光原位杂交(FISH,fluorescence in situ hybridization)和测序技术已能揭示许多不同尺度之下的三维基因组结构,然而这些方法往往需要将细胞固定,因此只能揭示静态的结构。
但是,除了静态结构之外,染色质的动态变化也至关重要,即需要在了解 DNA 三维结构的基础之上,通过增加第四个维度——时间,去观测染色质的组织结构随着时间的变化。比如,调控元件增强子(enhancer)是如何与远距离的特定靶向基因的启动子 (promoter) 相互作用的?这种动态相互作用又是如何调控基因转录的?然而,由于缺乏在活细胞中实时标记和追踪 DNA 的可靠方法,导致三维基因组的动态变化及其与基因表达的因果关系这一关键科学问题至今仍不明确。因此,发展一种能在活细胞中进行高时空分辨率的实时标记和追踪 DNA 的技术很有必要。
在活细胞中针对特定染色质区域进行成像需满足两个核心要求:首先,需要募集足够的荧光标记以便实现高信噪比(SNR,signal-to-noise ratio),同时还得确保染色质天然结构的完整性;其次,需要拥有较高的时空分辨率,以便捕捉染色质的动态变化过程。传统成像系统通过在基因组特定位点插入外源重复序列并利用 LacI-GFP 的序列特异性结合来进行成像。然而,该方法不仅需要耗时数月通过复杂的基因组编辑操作来构建稳定细胞系,同时其所引入的约为 10kb 的大片段外源序列还可能会干扰染色质的天然三维结构和功能。
所谓“他山之石,可以攻玉”, CRISPR 技术不仅是一种强大的基因编辑技术,也为 DNA 活细胞成像领域提供了革新性的解决方案。比如,可以将 CRISPR 系统中具有剪切 DNA 功能的 Cas9 改造成为核酸酶失活的 Cas9 蛋白(nuclease-dead Cas9)dCas9。尽管后者并不具备“剪刀功能”,但是得益于 sgRNA 的特异识别能力,让 dCas9-sgRNA 依然能像“邮差”一样,将荧光信号精准地递送至特定的基因位点从而可以用于成像。
通过将荧光标记系统与 sgRNA 的序列特异性识别能力加以结合,以及在 dCas9 的帮助之下,CRISPR 系统成功实现了活细胞染色质的实时动态成像。使用该技术的时候无需外源序列插入,只需通过设计特异性 sgRNA 即可精准定位目标基因组区域,在保持染色质天然状态的同时,还能显著提升操作便捷性。然而,绝大多数基于 CRIPSR 的 DNA 成像方法都仅适用于针对重复基因组区域进行成像。
由于增强子(enhancer)、启动子(promoter)等许多关键的基因组元件多为非重复序列,这一局限性极大限制了三维染色质组织结构的研究。同时,许多方法都依赖于调控多个质粒的表达, 或是需要构建稳定的细胞系,因此并不适用于与疾病有密切关系的原代细胞(即从人体分离的细胞),故而其应用范围受到了限制。
所以,亟需一种高时空分辨率的、适用于包括原代细胞在内的多种细胞类型的、能对包括重复 DNA 序列和非重复 DNA 序列进行活细胞成像的技术。
(来源:Cell)

和诺奖得主团队将论文发在 Cell
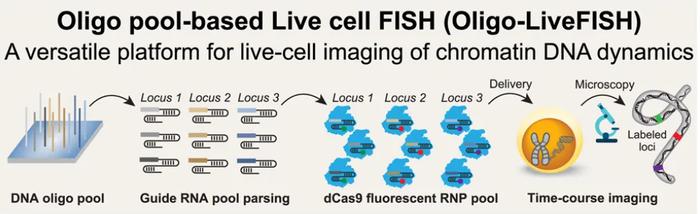
研究团队表示,Oligo-LiveFISH 能将计算机设计生成的靶向基因组位点的 DNA 寡核苷酸库(oligo pool)进行体外转录从而得到 crRNA 库或 sgRNA 库, 再通过化学方法引入荧光信号,进而以较低的成本得到能对非重复位点成像的 RNA 库, 再与 dCas9 蛋白在体外组装为 fRNPs,并通过电转的方法递送至细胞内进行成像。
其中涉及到十多个步骤,每一个步骤的参数都需要探索优化,包括反复优化最初设计生成 oligo pool 的程序以便确保剔除掉脱靶的探针。不过,总的来说,第一个步骤是要合成并标记上 gRNA,同时还得保证 RNA 不会被降解。为此,研究团队尝试了三种不同的标记方法,考虑到标记效率和可操作性,他们最终利用 poly(A)聚合酶和点击化学的标记方法进行高效荧光标记和纯化。
接着,还需要在显微镜下看到足够强的信号并能保证其特异性,同时还得(1)保证信号能被追踪一段时间而不会消失;(2)保证细胞处于健康状态;以及(3)保证追踪的轨迹不是由于诸如细胞运动等其他因素导致的。其中一个关键因素是:所有操作要尽量避免由 RNA 酶导致的 RNA 降解。“这让我们大约花费两年的时间去优化各种条件。”研究团队表示。
基于 U2OS 细胞将 Oligo-LiveFISH 平台搭建好以后,他们将其用于不同的基因位点和细胞种类。Oligo-LiveFISH 的独特优势之一在于无需构建稳定的细胞系,于是他们利用它实现了对于人原代 T 细胞和人胚胎干细胞诱导的神经元细胞的 DNA 活细胞成像并观察到了一些有意思的现象,比如 T 细胞中的“DNA doublets”。由于 T 细胞是悬浮细胞因此对其成像较为困难,而诱导的神经元细胞生存条件较为严苛,长时间的激光照射可能导致细胞死亡。为了针对这两种细胞进行活细胞成像和长时间追踪,并且保证细胞处于健康状态,研究团队又对其中许多步骤进行了修改和优化。
成功实现标记之后,还需提高追踪 DNA 位点时的空间分辨率和时间分辨率。研究团队与诺贝尔化学奖得主、斯坦福大学 W・E・莫纳(W.E.Moerner)教授合作,将 Oligo-LiveFISH 与超定位显微镜技术结合,以 20 纳米的高空间分辨率和 50 毫秒的时间分辨率追踪了非重复基因组区域。最初,他们使用的是二维超定位显微镜技术,后来团队使用了莫纳教授课题组开发的双螺旋点扩散函数(DHPSF,double-helix point spread function)显微镜实现了三维空间的超定位。该技术通过光学工程生成双光斑,利用两个光斑之间的角度代表轴向位置,从而能够以高时空分辨率实现同步追踪三维运动。研究团队发现染色质运动呈现高度 sub-diffusion 特性,符合分数布朗运动模型,这与之前利用插入外源的序列(如 LacO-LacI)的结果相符。
在平台搭建和优化好之后,他们测试了这一技术在两个方面的应用。一是研究了染色质结构和 DNA 通讯时间并提出了两种 DNA 动态相互作用模式:一维顺式通讯和三维反式通讯。此外,研究团队利用 Oligo-LiveFISH 技术研究了 FOS 基因转录与其增强子-启动子动态行为之间的关联。
总的来说,这项研究集合了细胞工程、CRIPSR、RNA 化学、三维显微镜技术、机器学习、物理建模等许多学科的知识和技术。
(来源:Cell)
日前,相关论文以《利用 Oligo-LiveFISH 进行染色质 DNA 通信的高分辨率动态成像》(High-resolution dynamic imaging of chromatin DNA communication using Oligo-LiveFISH)为题发在 Cell[1],朱彦宇是第一作者,斯坦福大学亓磊教授和莫纳教授担任共同通讯作者。

图 | 相关论文(来源:Cell)
未来,他们将用 Oligo-LiveFISH 来深入研究一系列的生物学问题。具体来说,会将 Oligo-LiveFISH 技术与 CRISPR 干扰/激活(CRISPRi/ CRISPRa)、表观遗传编辑等扰动技术相结合,用于解析增强子-启动子动态与转录调控及表观遗传修饰之间的因果关系。
参考资料:
1.Zhu, Y., Balaji, A., Han, M., Andronov L., Roy, A.R., Wang, H., Wei, Z., Chen, C., Miles, L., Cai, S., Gu, Z., Tes, A., Yu, B., Uenake, T., Lin, X., Spakowitz, A., Moerner, W. E., Qi, L. S., Robust High Resolution Dynamic Imaging of Chromatin DNA Communication using Oligo-LiveFISH. Cell. 2025,188,1-19
运营/排版:何晨龙